พบ ไวรัสก่อโควิด สายพันธุ์แอฟริกาใต้ B.1.351 ที่คลัสเตอร์ตากใบ นราธิวาส คาดอาจเป็นคลัสเตอร์ติดเชื้อต่อเนื่องในไทยจากผู้ลักลอบเข้าเมือง
เมื่อวันที่ 22 พ.ค. 64 ผู้สื่อข่าวรายงานว่า แฟนพจเฟซบุ๊ก Center for Medical Genomics หรือ ศูนย์จีโนมทางการแพทย์ รพ.รามาธิบดี มหาวิทยาลัยมหิดล ได้เผยแพร่ข้อมูลเกี่ยวกับการแพร่ระบาดโควิด-19 ดังนี้ ไวรัสก่อโรคโควิด-19 สายพันธุ์แอฟริกาใต้ B.1.351 มีการกลายพันธุ์ในตำแหน่งที่คาดว่า มีผลกระทบต่อการตอบสนองของระบบภูมิคุ้มกันมนุษย์ต่อไวรัส และลดประสิทธิภาพการทำงานของวัคซีน แต่มิได้แปลว่าวัคซีนจะใช้ไม่ได้ เพียงแต่ต้องเพิ่มอัตราส่วนประชากรผู้ได้รับวัคซีนให้สูงขึ้นเพื่อเกิดการป้องกันระดับประชากร
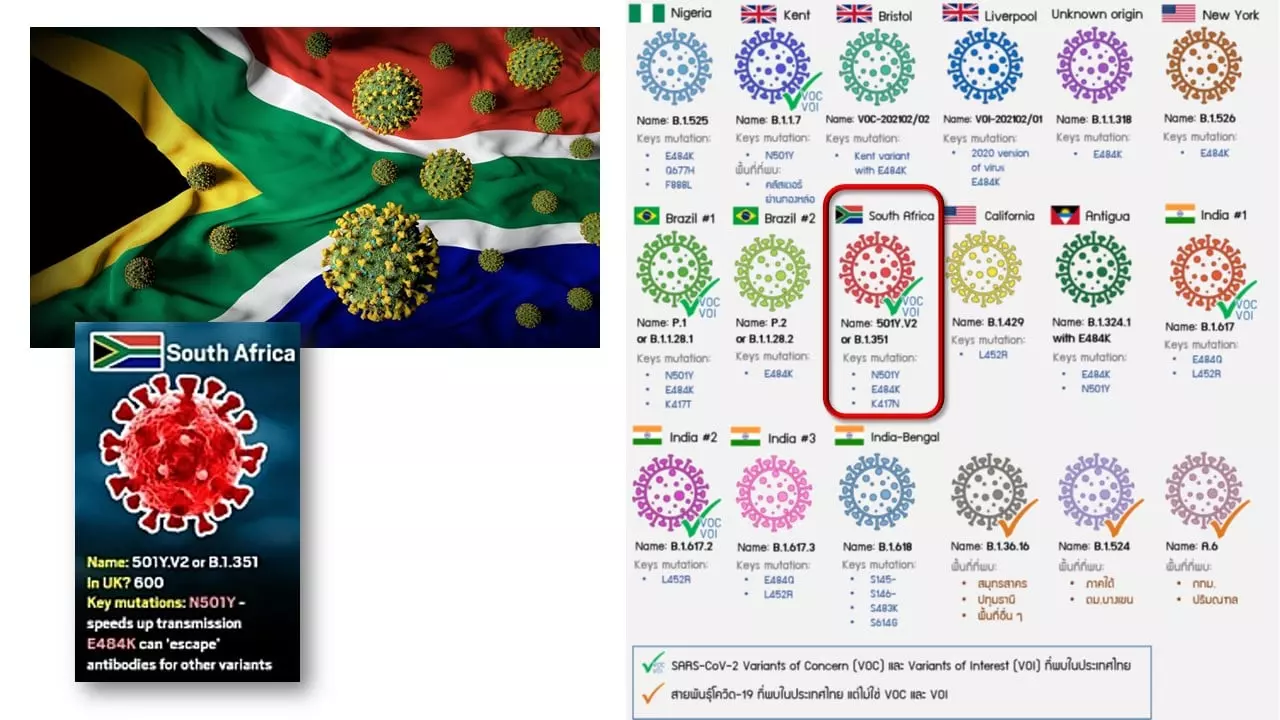
เชื้อไวรัสก่อโรคโควิด-19 B.1.351 หรือ 20H/501Y.V2 หรือที่เรียกกันติดปากว่าเชื้อโควิดกลายพันธุ์แอฟริกาใต้ พบครั้งแรกในอ่าวเนลสันแมนเดลา เขตปริมณฑลของจังหวัดอีสเทิร์นเคปของแอฟริกาใต้ในเดือนตุลาคม พ.ศ. 2563 รายงานโดยกระทรวงสาธารณสุขของประเทศแอฟริกาใต้เมื่อวันที่ 18 ธันวาคม พ.ศ. 2563 จากการวิเคราะห์ รหัสพันธุกรรมชี้ให้เห็นว่าไวรัสกลายพันธุ์ดังกล่าวเกิดขึ้นในบริเวณอ่าว Nelson Mandela แล้วตั้งแต่เดือนกรกฎาคม หรือสิงหาคม 2563
การระบาดในประเทศไทยของเชื้อโควิด-19 สายพันธุ์แอฟริกาใต้ B.1.351 (20H/501Y.V2) ณ วันที่ 22 พฤษภาคม 2564
...
ทางกลุ่มพันธมิตร COVID-19 Network Investigations (https://coni.team/) ได้รับการประสานจากทางกระทรวงสาธารณสุขให้ร่วมสืบสวนคลัสเตอร์ ที่อำเภอตากใบ จังหวัดนราธิวาส โดยได้รับข้อมูลว่าอาจเป็นคลัสเตอร์ติดเชื้อต่อเนื่องในประเทศไทยจากผู้ลักลอบเข้าเมือง
จากการถอดรหัสพันธุกรรมระดับจีโนมพบว่าเป็นเชื้อสายตระกูล B.1.351 ทางกลุ่มฯ ได้ส่งต่อข้อมูลทั้งหมดให้กับหน่วยงานของกระทรวงสาธารณสุขในระดับภูมิภาคและระดับชาติเพื่อสื่อสารกับประชาชนต่อไป ทั้งนี้ทางกลุ่มขอสื่อสารข้อมูลชุดนี้ให้ประชาคมวิทยาศาสตร์เพื่อร่วมกัน ติดตาม เฝ้าระวัง และพัฒนาวิธีป้องกันรักษาโรค โดยมีรายละเอียดดังนี้
1. ตัวอย่างชุดนี้จัดเก็บโดยทางกระทรวงสาธารณสุข เมื่อวันที่ 13 พ.ค. 2564 ทางกลุ่ม ฯ ได้รับตัวอย่างเมื่อวันที่ 17 พ.ค. 2564 และคัดเลือกมาสามตัวอย่าง (รหัส BKKCOVID720, BKKCOVID721 และ BKKCOVID722) เพื่อถอดรหัสพันธุกรรมระดับจีโนม โดยตำแหน่งที่ถอดได้มีลำดับการเปลี่ยนแปลงทางพันธุกรรมตรงกับสายตระกูล B.1.351 ตามระบบ PANGO แสดงใน (ภาพ 2)

2. การถอดรหัสใช้วิธี MinION ของ Oxford Nanopore Technologies (ภาพ 2) โดยมาจากวิธี amplicon sequencing ของ ARTIC Network ผ่านการวิเคราะห์โดย workflow ของ COG-UK และระบบ QC ของกลุ่ม ด้วยทรัพยากร Supercomputer จาก ThaiSC TARA
3. Genomic coverage ของสามตัวอย่างอยู่ที่ 85.01%, 90.11% และ 84.93% ตามลำดับ ปกติวิธีที่ใช้จะได้ coverage ประมาณ 95-98% แต่ตัวอย่างน่าจะมีการเสื่อมสลายระหว่างขนส่ง เพราะวิธีการอื่นที่ใช้วิเคราะห์ตัวอย่างนี้ก็มีปัญหาคล้ายคลึงกัน อย่างไรก็ดีตำแหน่งที่ได้เพียงพอต่อการกำหนดสายตระกูล ทางทีมถอดรหัสจะใช้วิธีการ Illumina เพื่อเพิ่ม coverage ของตัวอย่างชุดนี้ต่อไป
4. ทางกลุ่มจะนำข้อมูล BKKCOVID721 ที่ได้เข้าสู่ระบบ GISAID และจะรวมเข้าไปใน Nextstrain build รอบถัดไป พร้อมทั้งติดตามต้นตอการระบาดด้วยวิธี Maximum-likelihood และ Bayesian analyses
5. เชื้อสายตระกูล B.1.351 มีการกลายพันธุ์ในตำแหน่งที่คาดว่ามีผลกระทบต่อการตอบสนองของระบบภูมิคุ้มกันมนุษย์ต่อไวรัส และลดประสิทธิภาพการทำงานของวัคซีน แต่มิได้แปลว่าวัคซีนจะใช้ไม่ได้ เพียงแต่ต้องเพิ่มอัตราส่วนประชากรผู้ได้รับวัคซีนให้สูงขึ้นเพื่อเกิดการป้องกันระดับประชากร
ทางกลุ่ม CONI ประกอบด้วยนักวิทยาศาสตร์ที่สังกัดสถาบันในไทยและต่างประเทศ ทางกลุ่มทำการถอดรหัสโดยไม่เก็บค่าใช้จ่ายเพื่อช่วยหน่วยงานด้านการแพทย์และสาธารณสุขสืบสวนโรคและติดตามการระบาดในระดับประชากร
...
ข้อมูลจัดเตรียมโดย นายขจร จุลศักดิ์ AFRIMS, ผศ.ดร.ธนรรถ ชูขจร มหาวิทยาลัยมหิดล, พญ.ดร.ธีรรัตน์ คชการ Molecular Infection Medicine Sweden, นางสาวน้ำฝน โคตะนันท์ มหาวิทยาลัยมหิดล, นางสาว วิภาพร ทับทิมทอง มหาวิทยาลัยมหิดล, ดร.อลิซาเบธ แบตตี้ มหาวิทยาลัยมหิดล Oxford University, ดร.เอกวัฒน์ ผสมทรัพย์ มหาวิทยาลัยมหิดล, ดร.อินทรีย์ เสนสอน ศูนย์จีโนมทางการแพทย์ รพ.รามาธิบดี มหาวิทยาลัยมหิดล และศ.ดร.วสันต์ จันทราทิตย์ ศูนย์จีโนมทางการแพทย์ รพ.รามาธิบดี มหาวิทยาลัยมหิดล